
本文共 2099 字,大约阅读时间需要 6 分钟。


生物医学或其他研究论文中的“表一”多为基线特征的描述性统计。使用R单独进行统计,汇总,然后结果复制到excel表中,耗时耗力且易错!
tableone包“应运而生”,可以非常简单快捷的解决这个问题,重点是学习成本很低,大概几分钟?
一 载入数据 R包
## install.packages("tableone")library(tableone)library(survival)data(pbc)head(pbc) 
二 单组汇总
1 汇总整个数据集
对pbc整个数据集进行描述汇总,使用CreateTableOne()即可
tab1 <- CreateTableOne(data = pbc)print(tab1)
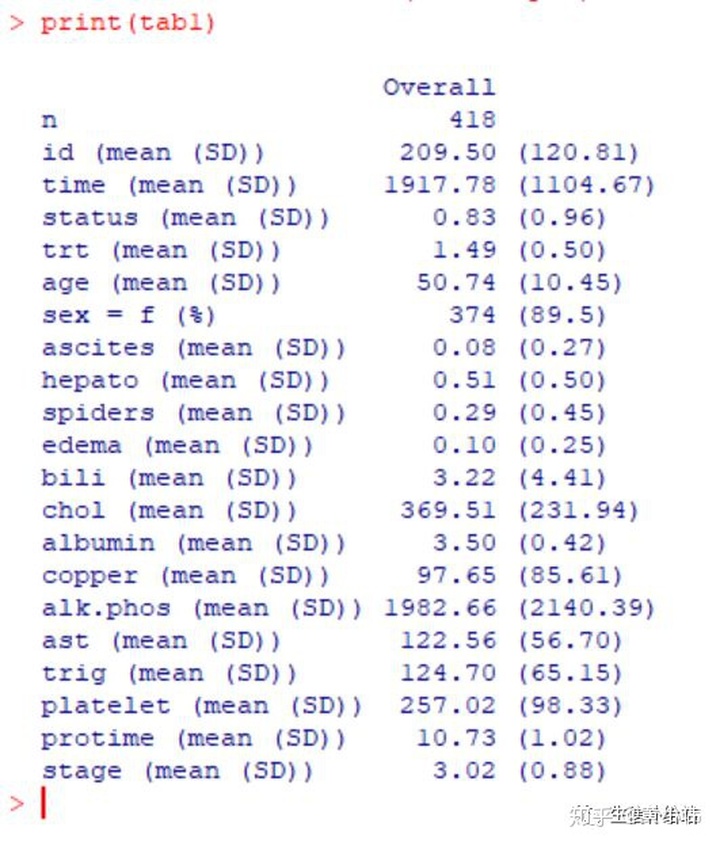
由于数据中的分类变量是数值形式,所以分类变量展示的也是均值(标准差)。
2 设置变量类型
dput(names(pbc)) # 输出据集变量名称## 需要汇总的变量myVars <- c("time", "status", "trt", "age", "sex", "ascites", "edema", "bili", "copper", "ast", "stage")## 需要转为分类变量的变量catVars <- c("status", "trt", "ascites", "stage")## Create a TableOne objecttab2 <- CreateTableOne(vars = myVars, data = pbc, factorVars = catVars)print(tab2, showAllLevels = TRUE) 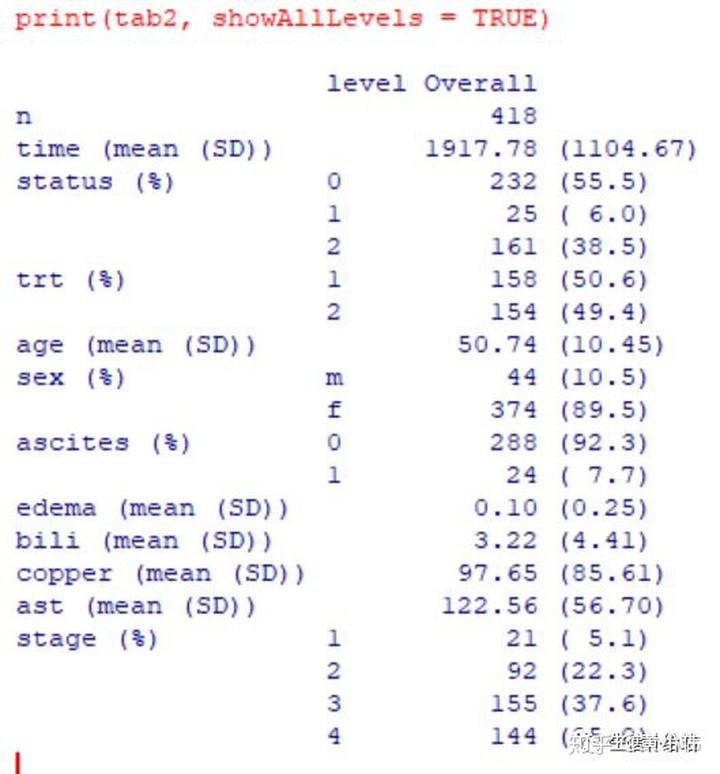
showAllLevels = TRUE 会展示分类变量的所有分类因子的结果。
此处随意选择一些变量进行功能展示, 分类变量显示计数和百分比 。
3 非正态分布变量
由于默认连续变量呈正态分布,因此上面的连续变量均表示为均数+标准差。
实际数据中的非正态分布数据,可通过nonnormal指定,则此变量展示为中位数(四分位数)。
#假设"bili","chol","copper"非正态分布biomarkers <- c("bili","copper","ast")print(tab2, nonnormal = biomarkers) 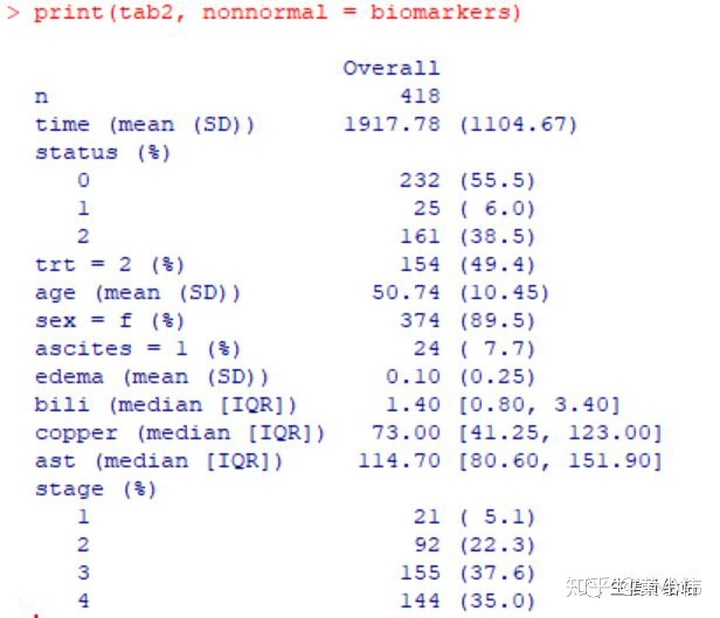
可见"bili","ast","copper"均用中位数(四分位数)表示;如果设置nonnormal = TRUE,则所有变量都按非正态分布处理。
三 多组汇总
1 分组统计
实际结果中,通常需要对数据集按照某个变量的分组进行汇总。下面展示使用trt进行分组汇总:
tab3 <- CreateTableOne(vars = myVars, strata = "trt" , data = pbc, factorVars = catVars)tab3
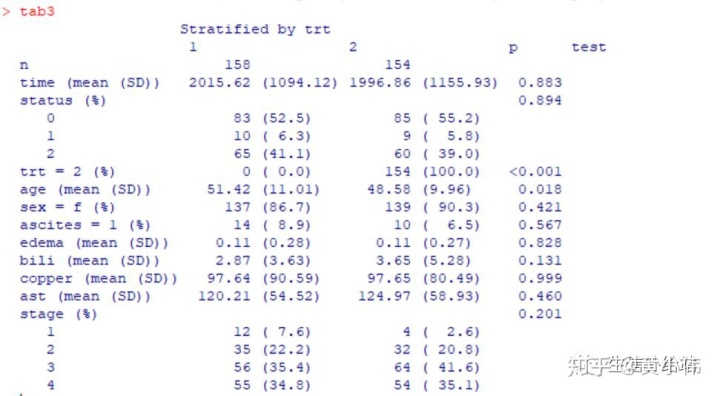
注意NA不作为分组
结果可看出,对trt进行分组且对每一组均进行了汇总,且统计输出了检验的P值。
检验方法:分类变量默认使用卡方检验 chisq.test();连续变量默认使用方差分析oneway.test(),当两组时方差分析等用于t检验。
2 定义检验方式
非正态性的数据展示方式为中位数(四分位数),检验方式也最好不使用T检验:
非正态分布的连续变量使用kruskal.test()检验,两组间比较时,kruskal.test()和wilcox.test()等效;
分类变量可使用fisher.test()进行fisher精确检验,通过exact()指定进行fisher精确检验的变量。
#addOverall 添加Overall信息tab4 <- CreateTableOne(vars = myVars, strata = "trt" , data = pbc, factorVars = catVars, addOverall = TRUE )#exact设置fisher精确检验的变量print(tab4, nonnormal = biomarkers, exact = "stage")

四 导出结果
使用write.csv一键导出结果
tab4Mat <- print(tab4, nonnormal = biomarkers, exact = "stage", quote = FALSE, noSpaces = TRUE, printToggle = FALSE, showAllLevels = TRUE)## 保存为 CSV 格式文件write.csv(tab4Mat, file = "myTable.csv")
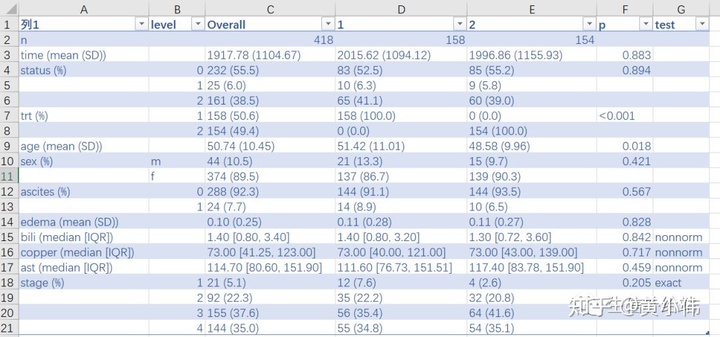
随便套用了一个表格格式,可以在excel中弄成喜(文)欢(章)的样式,这个自己发挥吧。
参考资料:
https://cran.r-project.org/web/packages/tableone/vignettes/introduction.html
转载地址:https://blog.csdn.net/weixin_34081553/article/details/112702569 如侵犯您的版权,请留言回复原文章的地址,我们会给您删除此文章,给您带来不便请您谅解!
发表评论
最新留言
关于作者
